如何挖掘微生物抗性基因?
抗生素药物的大规模使用促使大量新耐药菌株的产生。新型耐药微生物影响着人类健康,农业养殖等多个方面,并在水体、土壤、活性淤泥、动物体内都已被发现和报道。耐药菌株的发展令疾病治疗的难度和成本提高,因此对它们的研究也极受关注。
耐药微生物研究最为核心的部分就是耐药基因的鉴定与挖掘。通过对耐药基因的分析,我们可以了解其进化发展历史,也可以解开其耐药机制,为后续药物开发以及疾病治疗提供重要参考资料。
现阶段,通过抗性基因数据库对耐药基因的鉴定挖掘是常用手段。常用的抗性基因数据库有ARDB,AGRO,BacMet,APD3,CARD,ARG-ANNOT等。但由于AGRO只是有抗万古霉素和β-内酰胺两类抗生素的基因信息,BacMet则专注于杀菌剂和金属抗性基因,而APD3则关注抗菌多肽,因此我们今天主要讨论两个比较广泛的数据库:ARDB和CARD。
ARDB数据库[1]
在线网址:https://ardb.cbcb.umd.edu/
引用文章:从2009年开始已经有400多的引用文章,这对一个专业性数据库来说是个不错的数据。

数据库介绍
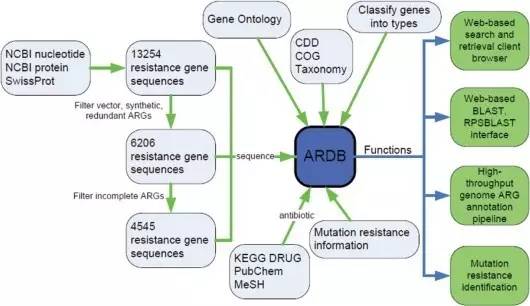
ARDB(Antibiotic Resistance Genes Database)数据库主要包含细菌病原菌的多种抗性基因数据。在08年刚建库时候只收集了13254条抗性基因序列,通过去除质粒、人工合成、冗余以及不完整序列之后,最后整理了4545条核心信息序列。到09年7月左右,数据库最终扩增到23137条序列(未筛选)。然后结合GO、KEGG等信息,通过该数据库的注释,可以找到耐药性相关基因的名称,所耐受的抗生素种类等信息。
ARDB数据库的核心功能主要分为:基因信息查询、序列比对注释以及同源序列对比。其中序列比对注释是通过BLAST来完成,具体操作见“使用说明”部分。
展开全文
ARDB建库原理图
使用说明
1
打开网址
https://ardb.cbcb.umd.edu/
2
基因信息查询
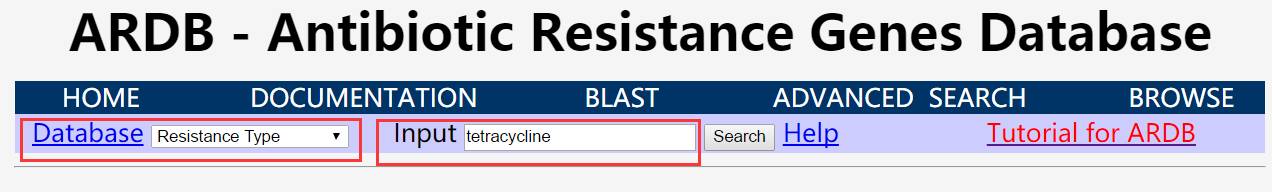
如果疾病研究或克隆需要,要了解某类型微生物的抗性情况,可以选择在线搜索功能。打开网页,在下图中第一个条目选择要搜索的类别:如抗性类型、抗性物种等。然后在第二个条目中输入关键词信息,点击搜索,就可以找到对应抗性基因信息。如下图,第一列选择抗性类型,第二列则填写tetracycline这一类抗生素信息,就可以出来有关tetracycline的抗性基因(如tetR)。
3
序列比对注释
如下图所示,点击BLAST,就会跳转出下面的画面。如果有已知蛋白或DNA序列,想知道此序列是否为抗性基因,则可以通过“Regular BLAST”比对获得序列抗性信息。如果通过基因组测序或宏基因组测序拼接获得基因组后,想知道基因组中是否有抗性基因,则可以选择“Genome Annotation”进行比对。

4
同源序列对比
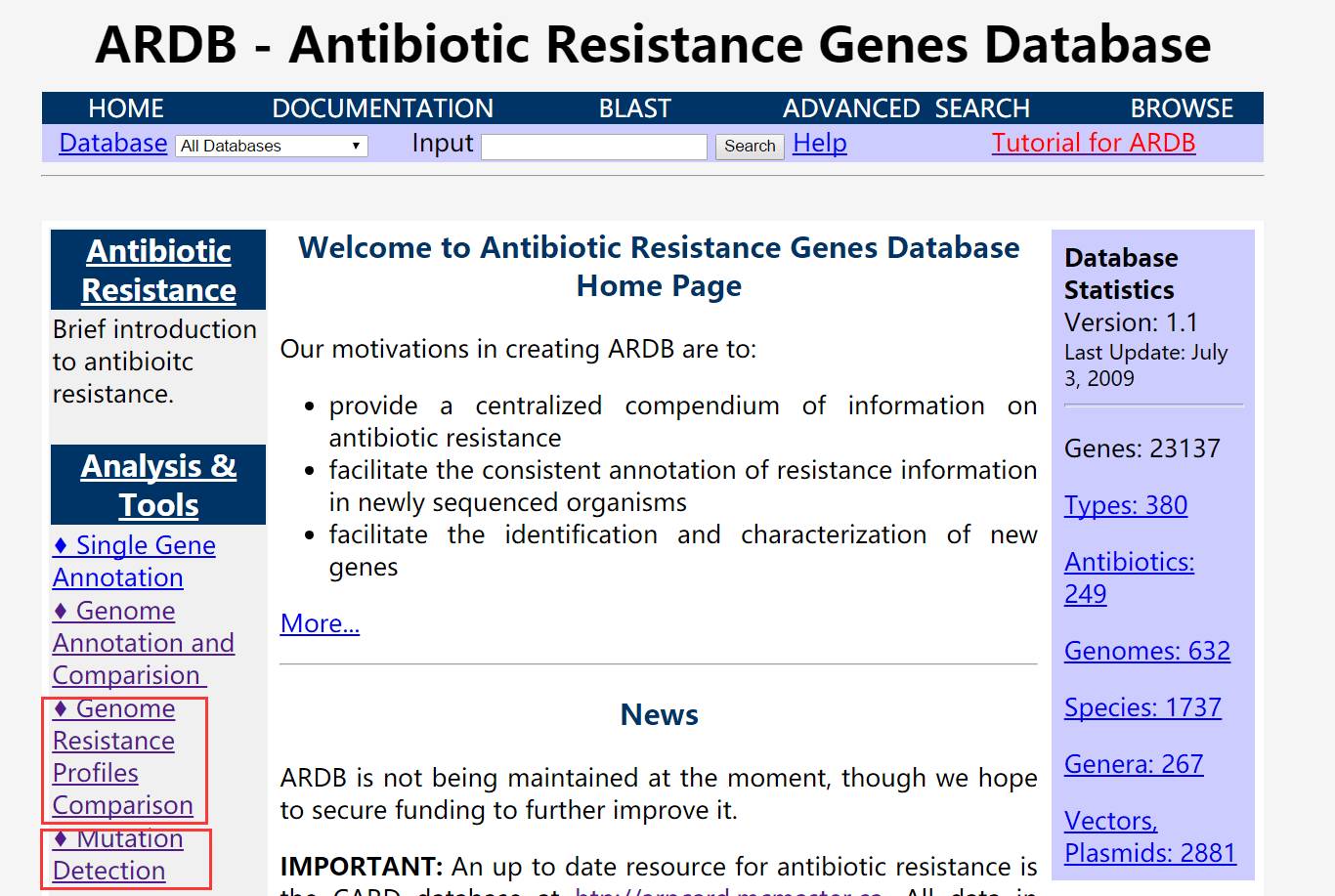
抗性基因的突变会影响抗性效果,因此很多研究需要研究抗性基因的变化情况,这可以通过序列之间的对比完成。如果需要了解某类基因的碱基变异情况,只要选择下图中的“mutation detection”,上传序列信息,选择数据库提供16S rRNA、23S rRNA、gyrA、gyrB、parC、parE、rpoB、katG、pncA、embB、folP、dfr等12类基因的参考序列,即可完成比较,挖据基因突变信息。同时,用户如果有新的测序物种,也可以选择下图的“Resistance Profiles Comparisons”,即在全基因水平与数据库的已知物种进行抗性基因比较,在物种水平完成抗性基因进化研究。
CARD数据库[2][3]
细心的朋友会发现,ARDB(Comprehensive Antibiotic Resistance Database)数据库虽然综合性强,引用率高,但它在2009年开始已经没有更新了。这对日新月异的抗性基因研究来说是一件极其受限制的事情。因此,有没有一个更好的耐药基因数据库呢?其实答案就在ARDB的官网当中,它们推荐了CARD这个数据库(见下图)。

数据库介绍
上一篇:宏基因组学在微生物抗生素抗性基因检测中的应
下一篇:大型干货现场!抗生素抗性基因(ARGs)分析一站