【生物信息学笔记】UCSC基因组浏览器
这个Genome Browser也是我们用的非常多的一个资源。它也是有网上的这样一个界面。它的这个特点是所包含的这些物种,都是以一个基因组的坐标为框架。
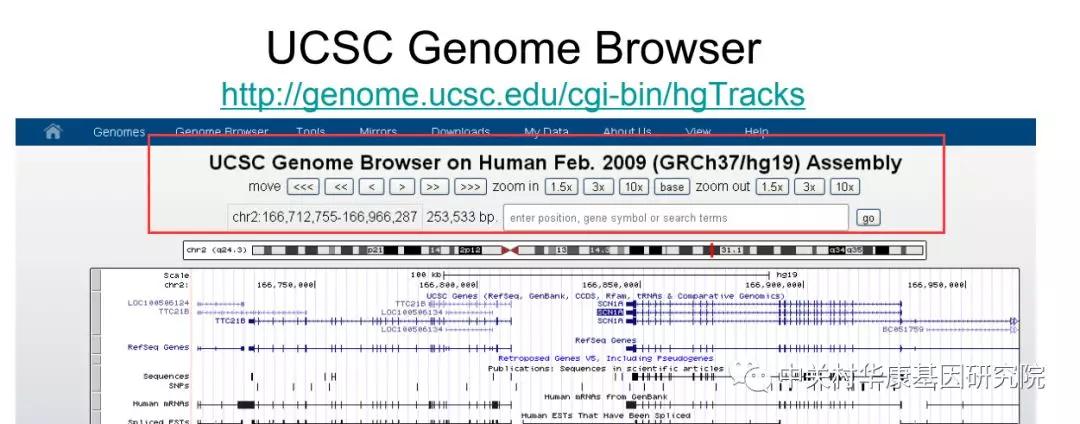
然后,你看这个就是它的基因组的坐标。这个就是2号染色体从这里到这里这样的一个坐标。每一类数据都是横着的这样叫做一个track。
那比如说这有RefSeq对于这基因的注释,那这个里边高的竖道就是一个外显子,中间的一个横杠就是它对应的这个内含子的区间。空白的就是基因间的区域。
那下边就有:落在这个序列上有哪些SNP,有哪些已知的mRNA,有哪些剪切的EST; 它还有一个track就是没有剪切的EST,质量就会要稍微弱一点,但是也是有它的意义的。 有哪些是被高通量实验测量出来的DNase的cluster,有哪些是tranion factor,通过ChIP-Seq鉴定出来的tranion factor的binding sites。
展开全文
然后上图圈出来的这一个地方就会显示你感兴趣的这个物种的近缘种中这个片段[的相似序列]有还是没有,保守性有多强。 比如说对于人的一段基因,它就有从rhesus monkey到mouse、dog、elephant等等很多的近缘物种的相关的基因。
下边还有就是它有哪些是repeat、repetitive element。这只是显示了其中的一小部分。
这个上面显示的是能让你可以在基因组左右滑动, 你可以滑动一点或滑动很大的规模,并且你可以zoom in或zoom out,就是你是想看一个大的尺度还是一个小的尺度。 然后你可以搜索,你可以用坐标搜索,用基因名搜索,等等。
这里显示的是一个基因结构。你看到这里有箭头,箭头显示的是它是正链还是反链上的基因。因为在人的基因组,40%的区域的正链和反链上都有转录信号。
虽然人的基因组真正编码蛋白的基因只有两万多,但其实转录信号覆盖了基因组的绝大多数的位置,而且是正链反链经常都有转录本。 你通过UCSC[ Genome Browser]可以看到这些转录的信号。
它还有一些track是表观遗传学的实验鉴定出来的信息,包括DNA甲基化,组蛋白修饰,DNase Hypersensitivity region,ChIP-Seq鉴定出来的转录因子结合位点等等。
另外不同物种之间的保守性也是很直观地就可以看到。所以你[如果有]感兴趣的基因或蛋白, 一定要到UCSC[ Genome] Browser上去看一下,看看已经有了哪些已知的信息。
对于遗传变异,也有很多的track来描述。 如果是一个和疾病相关的基因,有哪些和疾病相关的信息,也有不同的track来显示。
比如说,如果是一个和癌症相关的基因,它有已知的体细胞突变的话, 那只要[这个突变]在COSMIC数据库被包含,那[这个突变]在UC Santa Cruz的Genome Browser上也可以看到。
UCSC有一个非常有用的工具,你可以在网上[用],也可以把它打包下载下来、在本地把它嵌入到自己的程序里;它就是BLAT。
BLAT和BLAST不太一样的地方是,它主要是[用于把]一个基因或蛋白的序列比对到它自己所在的物种的基因组序列上。 而BLAST则经常是做跨物种的比较,所以BLAST最后找到的结果可能是另外一个物种的和你这个序列相似性只有20%、30%序列一致性的结果。 而BLAT做的事情是,它最后真的能比对上的[位置]就是它这个基因本来应该从基因组的哪里出现。 你想象一下,如果你所有实验都是100%准确的话,那你的序列和基因组上的这一段片段应该是100%吻合的。 但事实上,你很少看到100%吻合,原因第一是测序经常有错误,第二是你的样本或所研究的个体,它和reference genome[相比]本来就是有遗传变异的。 所以这些差异,不管是本来样本之间的差异还是测序错误造成的差异,你都需要在写程序的时候给它相应的处理。 所以BLAT就是对于这方面处理的很好。
另外UC Santa Cruz有一个很有用的、对大家应该都比较有用的工具:In-Silico PCR。
上一篇:CRISPR基因编辑会普遍增强具有癌变潜力的细胞
下一篇:如何选择/构建质粒载体