FPKM / RPKM与TPM哪个用来筛选差异表达基因更准确?
最近网络上充斥着关于FPKM / RPKM与TPM的帖子,显然TPM越来越流行了,但很多测序公司的分析团队还没反应过来,仍然在麻木地把FPKM提供给客户。所以弄得很多小伙伴内心十分纠结,它们差异有多大?公司给我的是FPKM,对差异基因筛选有没有影响?

呵呵,今天你来对了,本文专治各种纠结!
1. RPKM / FPKM与TPM的差异
前文已经说了,这个问题搜一把可以搜出一大把,我就不说了。

但,你可能比我还懒,我还是简单贴一下概念吧!
RPKM:Reads PerKilobase Per Million Reads
FPKM:Fragments Per Kilobase Per Million Reads
TPM:Transcripts Per Million Reads
可以看出,RPKM与FPKM根本就是一回事,只是前者用的是单端测序(所以称为Read),后者用的是双端测序(双端测的是同一Fragment)。但现在基本上全是双端测序了,所以RPKM早已辉煌不在了。
TPM并不是最近出来的,只不过流行的比较慢,但最近越来越多的分析人员开始接受它,许多分析软件里的新星也都选择了它。
为什么?这当然是有原因的!
因为,TPM更好!
有一个重要指标可以很好说明这个问题:FPKM的创造者Pachter已经更倾向于使用TPM了!
在同一个样本内,无论是用FPKM还是TPM都是一样的,它们的相关性是1,TPM对总的测序reads进一步做了均一化,所以它们的最直观的差别就是,不同样本间TPM的总和是相同的,而FPKM的总和不同!可以想象,TPM使得我们更容易去比较同一个基因在不同样本中所占的read数的比例。
哦!TPM更好?那我更纠结了!公司只给了我FPKM啊,这对差异基因筛选有没有影响?我筛选出的差异基因很少是不是和这个有关?
2. 主流差异表达分析软件从来不用TPM作为输入
鉴于TPM的流行势头,下文方中仅以TPM代表所有RPKM / FPKM或TPM等此类均一化表达数据。
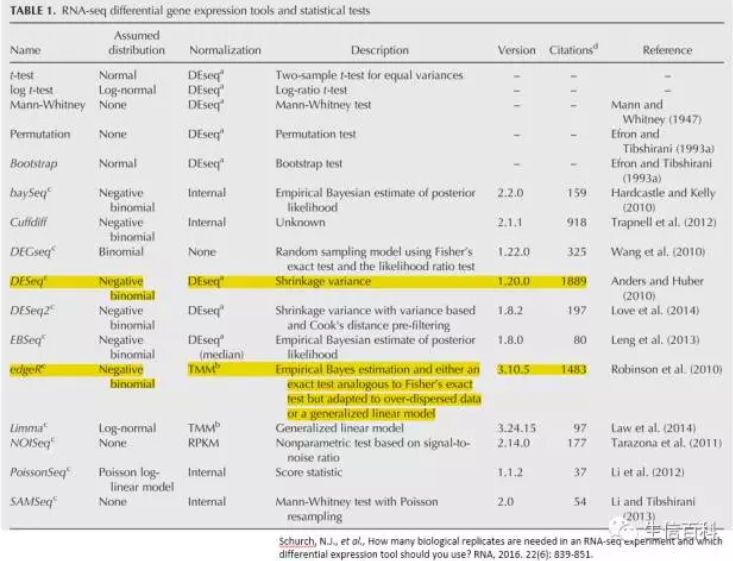
大部分差异表达分析软件并不支持使用TPM进行差异表达基因筛选(当然也有极少数不推荐的软件例外,如Tuxedo),最主流的DE(differential expression)分析软件DESeq2 和 edgeR ,包括我强烈向大家推荐的sleuth包,都强调输入数据为:Raw read counts (not normalized) !
为什么说它们是最主流的?并且我也只推荐这几种工具(推荐顺序sleuth > DESeq2 > edgeR),是因为它们经受了更多的测试,并且也是引用人数最多的,空口无凭,当然有引文数据为证:
上一篇:专家呼吁:应有条件允许开展细胞治疗研究
下一篇:新标准、新奖章!今年全国五一劳动奖的奖励有