Snakemake:简单好用的生信流程搭建工具
Snakemake是什么?
大多数生物信息初学者最早接触的生物信息分析流程,大多数是用脚本语言(Perl、shell、R、Python等)将生物信息流程中的各个功能写成一个个脚本,再用大脚本套小脚本的方式将整个流程打包封装。这种方式对编程能力有很高的要求,否则很容易出现流程bug、无法监控任务运行状态、无法有效查看任务日志等问题。等实验室终于出了个大牛将这些问题都解决了,可能大牛又要毕业了,新来的生信小白又只能看着层层叠叠的嵌套脚本“望洋兴叹”……
这时候就需要 Snakemake 闪亮登场了,因为snakemake可能是处理这类问题最简单、有效的一个方法了。比如将流程像下图这样搭建起来,清晰明了:
PS:这张流程图也是 snakemake 通过命令自动生成的奥,具体命令后面会有介绍。
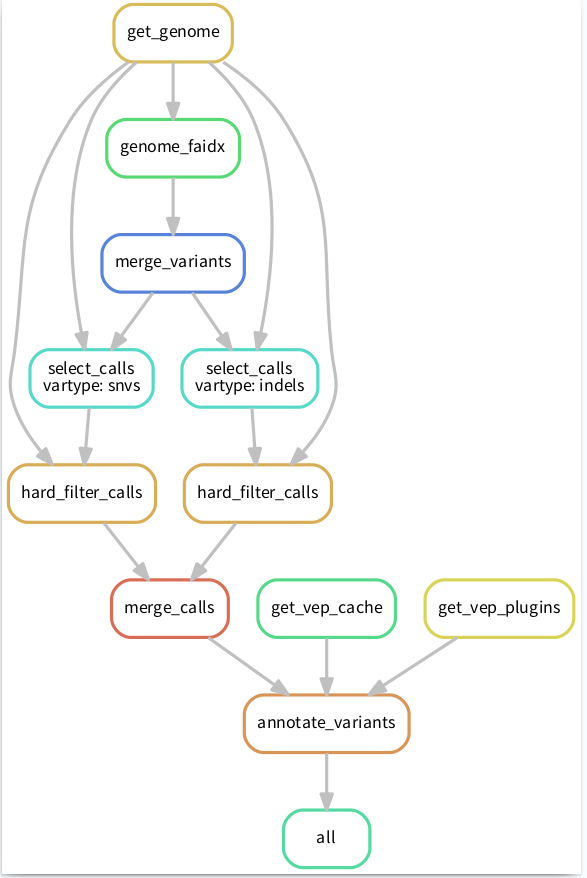
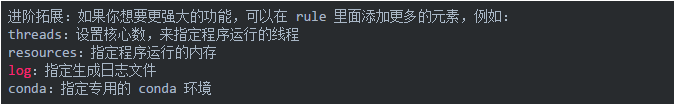
Snakemake 是基于 Python 的一款工具,所以它也继承了 Python 语言简单易读、逻辑清晰、便于维护的特点,同时它还支持 Python 语法,非常适合新手用户。snakemake 的基本组成单位叫“规则”,即 rule;每个 rule 里面又有多个元素(input、output、run等)。它的执行逻辑就是将各个 rule 利用 input/output 连接起来,形成一个完整的工作流,当检测到 input,就执行相应 rule;检测到 output,就跳过相应rule,根据这一规则,snakemake 还可以实现断点续投。
本教程全程在Linux环境下完成,为了顺利完成教程内容,请准备一个Linux环境。如果是windows用户,可以安装一个虚拟机;如果是win10用户,可以使用win10自带的子系统(WSL)安装Linux系统。
02软件安装这里建议使用conda安装软件,可以轻松解决各种依赖问题。同时建议将本教程中用到的软件安装在一个新的conda环境中,以避免不同版本软件间的依赖冲突。由于篇幅有限,conda的安装方法不再赘述。
该命令创建了一个名为snakemake_env的conda环境,并在该环境中安装了 snakemake 。
进入 snakemake_env 环境,查看上面要安装的软件,可以发现都已将安装完成。
如果你的conda安装很慢或者获取不了最新版本的软件,可以使用下面的命令安装:
03数据准备我们直接下载官网提供的测试数据:
解压后得到一个 data 文件夹,目录如下:


PS:建议将工作流文件命名为 py 文件,这样你在写 Python 代码时会有语法高亮奥。
上一篇:夏日“衣”触即发,将美好的自然色彩穿在身上
下一篇:山东烟台病例基因分型为奥密克戎BA.2.3进化分支