利用限制酶作图法构建物理图谱(PhysicsMap)
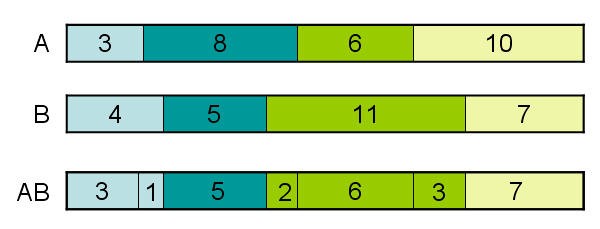
图1 酶切DNA片段示意图
在得知以上信息的情况下,如何对这些片段进行排序使得它们的顺序与实验结果一致呢?
对于这个具体例子,一个解决办法如图1所示。
双重消化的一种变化形式是部分消化(parital digest),这时只采用—种限制酶,但进行多次实验来消化同一目标分子的许多拷贝,得到作用于不向拷贝的不同的反应时间。,反应时间短,反应不完全,只有部分位点被限制梅切开,改变反应时间,就会得到不同的限制酶切割组合。反应时间的长或短决定了被识别的限制位点的多或少,因而产个不同长度的片段。理想情况下,这种实验对每一限制位点至少可提供一个片段,接着就可以通过分忻片段的长度来定位限制位点,如图所示,酶A可识别3个限制位点,因此该酶的部分消化实验就可以产生如下长度的片段集合:3、11、17和27(左端点与所有其他识别位点之间的片段,如11长度单位的片段是由于3和8之间的位点未被酶切产生的);8、14和24;6和16;16。
消化数据可能会有以下几种实验错误。首先,是片段长度测定的不确定性、片段的长度通常通过凝胶电泳来检测,因而其误差率可达5%;其次,如果片段太小的话,我们根本不可能检测它的长度;第三,有些片段还可能在消化过程中丢失了,这就导致了DNA分子覆盖之间存在空袭。所有这些错误都使作图过程变得更加复杂。即使这些问题并不存在,其有关的计算问题也相当困难。
限制酶作图的规模受限于限制片段的大小
如果使用的限制酶在DNA上切点相对较少,限制酶作图就比较容易。但如果切点较多,
作图时所需要测定的片段大小和需要比较的单酶消化、双酶消化和部分消化片段的数量也会增加。即使引入计算机分析,问题仍然存在。当消化产物中含有的片段多到一定程度时,琼脂糖凝胶中一些单一条带会重叠到一起,使一个或多个片段被错误检测或完全遗漏的可能性大大增加。即使所有的片段都能够被确定,如果存在大小相似的片段,也不可能将它们组成一个清晰的图谱。
因此,限制酶作图更适用于小分子,其长度依赖于靶分子中限制位点出现的频率。实际应用中,如果DNA分子小于50kb,通常可以选用6核苷酸识别序列的限制酶来作图。50kb一般低于细菌或真核染色体的下限,但一些病毒和细胞器的基因组位于这一范围内。在对这些小分子进行测序时,它们的全基因组限制酶图谱确实发挥重要的指导作用。细菌或真核巴组DNA被克隆后,其片段通常小于50kb,此时限制酶作图也同样有用。这表明构建详细的限制酶图谱可以作为对克隆序列测序的前提步骤。这是限制酶作用在大的基因组测序计划中的重要应用。但能否利用限制酶分析对大于50kb的完整基因组进行更全面的作图呢?
一些在靶DNA分子内具有罕见切点的酶,可以稍微克服限制性作图的局限性。这些稀有酶一般分为两类:一是具有7或8个核苷酸识别序列的酶;一是识别序列包含靶DNA中稀少基序的酶。
上一篇:基于质心查找的栅格检测镜像消除方法及装置与
下一篇:青白江中试之窗丨抢抓机遇打造“一带一路”中试产业基地