科学网利用Oligo7设计引物
因为这段时间实验的需要,总算有机会好好研究一下引物设计的方法,虽然有在网上找了一些课程,但是受到英语和其他相关水平的局限,总有些地方不是能看的太懂,所以就选择一种自己能够熟练掌握的方式,基本能满足自己的要求。写下来,算总结,也算分享,万一有人觉得还可以,我也挺高兴。关于引物设计的软件,有Oligo,DNAMAN等等,但是我目前只应用到Oligo。
使用情况一:将目的片段导入新的载体中。
对于Oligo的操作,首先通过File--New Sequence在打开的框中输入DNA模板序列,或者点击open直接添加文件,再点击√就可以了。(或者可以直接利用快捷键Ctrl+C/V进行复制粘贴)。
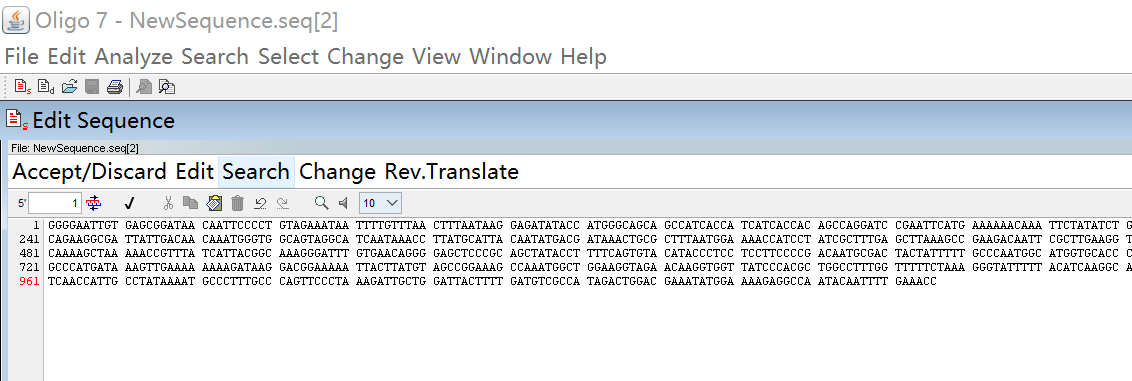
引物一般由保护碱基、酶切位点和与模板的互补序列三部分组成,长度一般在18~30bp。
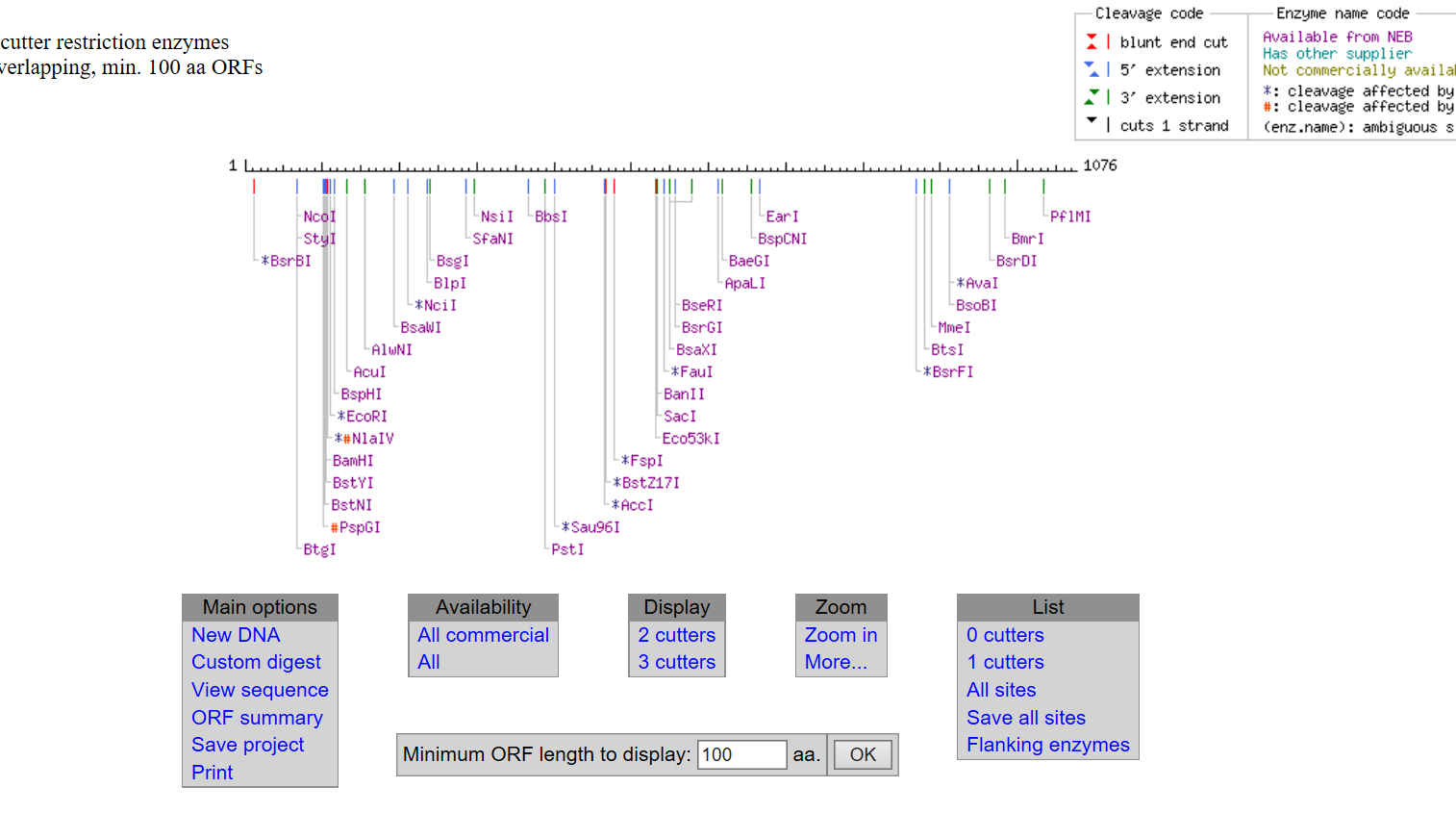
1.1 酶切位点的选择,我们要确保目的片段与载体上有且仅有一个该酶切位点。我常用的方法是利用NEBcutter V2.0,将目的片段的碱基序列输入框中(我常用的是Linear),选NEB enzymes,然后提交,会出现该目的片段上所有存在的酶切位点分析,选 0 cutters,即序列上不存在的酶切位点,再将搜索结果与待连接载体的MCS区域相比对,找到两个合适的酶切位点就可以了,(注意,所选的酶切位点不能靠的太近,最好不选相邻的酶切位点,他们之间的距离最好至少在几十个碱基以上,可以选择实验室常用的一些酶)。
1.2 保护碱基的选择,可以附件NEB保护碱基表, 在表格中找到相应的酶进行选择,优先选择裂解率高的保护碱基。
NEB保护碱基表.doc
1.3 与模板互补的片段,引物中需要有一小段片段与模板相结合,才能保证PCR过程的进行。可以先选择15bp左右,后期可进行调节修改。
由于有点时候没办法单纯靠想象记住这些序列,所以我常会在纸上将这一部分写出来分析,更加的直观。方法虽然比较笨,但是很有用,尤其是反向引物的设计,因为复制方向是5’-3’,需要的是反向互补序列,这个时候更容易出错。
选择Edit-Forward Primer,将初步设计的引物输入,点√确定。引物会自动对应到模板的位置上。注意复制进行的方向,不要设计反了。
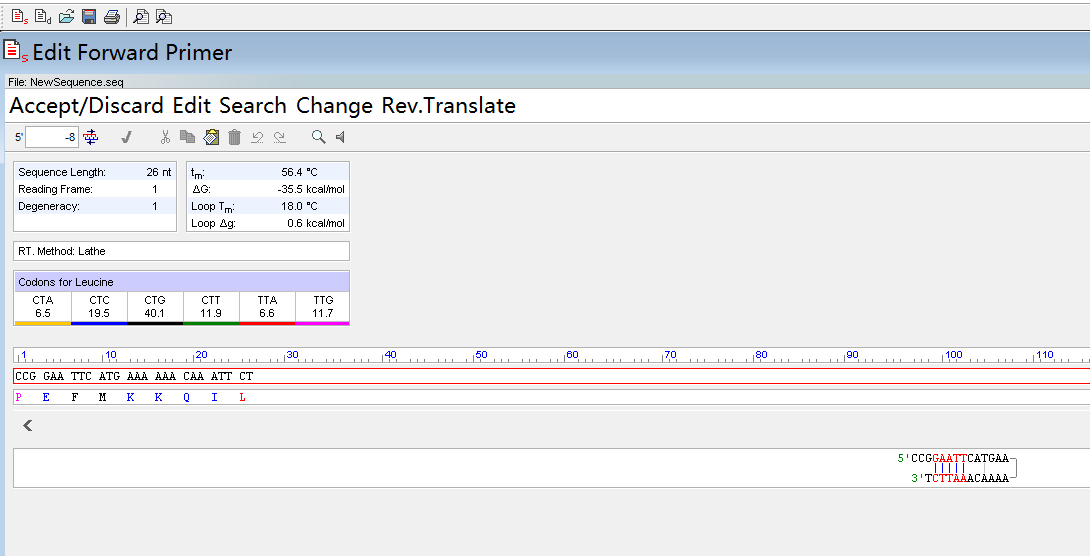
下游引物的设计原则和上游引物一样,但是所需的是互补序列,选择Edit--Reverse Primer进行输入就好了。
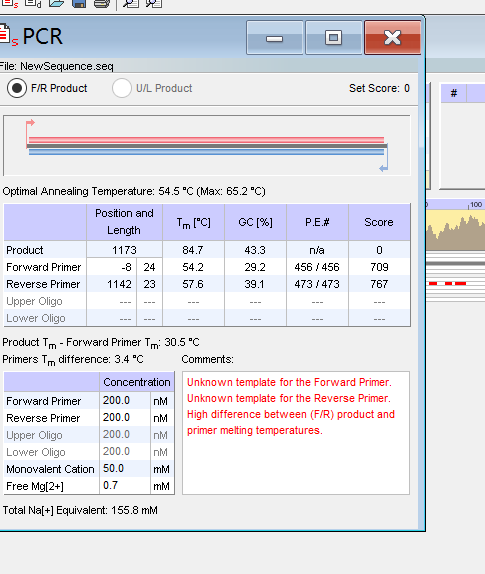
初步引物设计好之后需要进行分析来验证引物的有效性。这一部分主要在Analyze里面进行验证,主要包括引物二聚体的形成,发夹结构的形成,G/C含量,错配情况及同源性等方面。
2.1 引物的3’端最好保证是游离状态,有助于产物的伸长延伸,而5’端没有这个要求。
2.2 引物自身或引物之间不能带有连续4个以上碱基的互补,引物二聚体及发夹结构可能会导致PCR反应不能正常的进行;
2.3 引物的G+C含量在40%~60%之间,过高或过低都不利于引发反应;
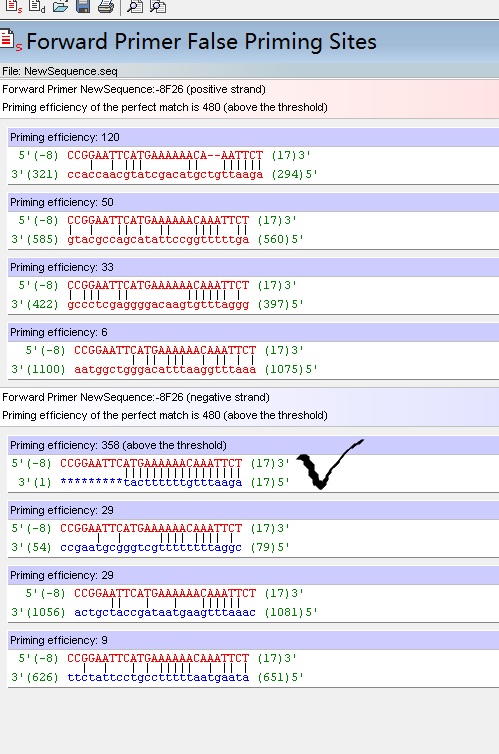
2.4 错配情况及同源性中,正确的产物对应的数据应该是最高的(above the threshold),这样才能保证正确产物的优胜表达。
2.5 上下游引物的Tm值差值不应超过5℃,退火温度按较低的Tm值选定。
在分别验证完之后可以选择Analyze-PCR进行模拟PCR,可以根据提出的建议进行修改。
使用情况二:敲除一小段基因片段。
Oligo的使用情况与上面所述相同,但是引物的组成上没有另外附加的酶切位点和保护碱基,此时引物分为两部分,前半部分与待切去片段前面的那一段序列互补,后半部分与待切去片段后面一部分互补。(参考:下图中被切去片段为118~180bp部分)
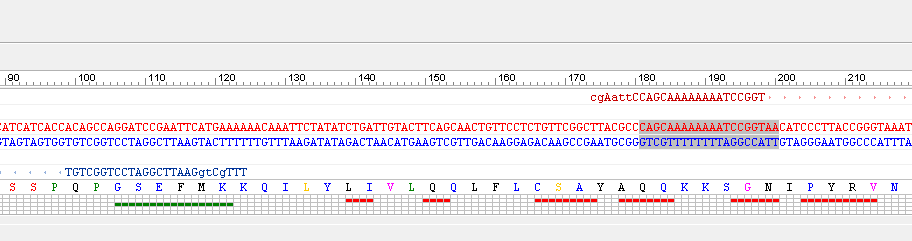
切去基因片段时应注意:
上一篇:功能超强大的免费肿瘤数据库
下一篇:国务院办公厅关于印发生产者责任延伸制度推行