食源性致病微生物全基因组测序进展汇总
近日,国家食品安全风险评估中心官网发布国家食源性致病微生物全基因组测序数据库及测序分析平台建设进展。

国际进展
微生物全基因组测序技术已经成为国际上食源性致病菌鉴定,分型及溯源的金标准技术
由微生物污染引起的食源性危害是中国目前的主要食品安全问题,也是世界各国面临的共同挑战。在应对各种致病微生物引起的食源性疾病暴发事件中,快速、准确溯源引起疾病暴发的微生物致病性食品非常重要。传统的食源性疾病暴发病原的确认通常需要几周或更长时间,全基因测序技术可在几天内确定病原,为及时控制并召回病因食品、有效诊治患者提供强有力技术支持。因此全基因测序技术已经成为国际上食源性致病菌鉴定,分型及溯源的金标准技术。
脉冲场凝胶电泳分析(Pulsed Field Gel Electrophoresis,PFGE),也称之为PFGE分子分型(致病菌确认)技术,是我国目前在核酸和分子水平上鉴定细菌的主要手段。PFGE分子分型技术始建于1996年,美国的50个州、美国FDA,美国CDC及美国农业部食品安全检测局(USDA/FSIS)均为其成员。PFGE分子分型技术对病原菌的DNA进行分型,称之为DNA指纹图谱(DNA fingerprinting),该技术可以在DNA水平鉴定和区分常见的食源性致病菌。全基因测序技术的迅速实用化,预计美国、欧盟在未来5年内将采用全基因测序技术替代目前传统的食源性致病菌鉴定,分型及溯源技术。
2008年美国FDA启动了国家微生物全基因组计划,2012年美国FDA和美国CDC宣布启动“10万基因组计划”,该项目历时五年,旨在合作创建一个包含10万种食源性致病菌基因组的公共数据库,以加速食源性疾病暴发的细菌鉴定。该计划将对约10万种重要的食品来源的致病菌(包括沙门氏菌、李斯特菌和大肠杆菌等重要的致病菌)测序,并将测序信息序储存在美国国立卫生研究院下属的国家生物技术信息中心(NCBI)的公共数据库。该数据库将开放访问,提供能鉴定病原体及其来源的检测方法,可以快速准确辨识致病食品。
2014年美国CDC启动了基于全基因组测序技术的高级生物学检测项目,宣布在2018年将不再使用PFGE分子分型技术,采用全基因组测序技术用于单增李斯特氏菌的分子分型,以便快速准确鉴定可疑的致病食品。2015年欧盟食品安全局(EFSA)已经将全基因组测序技术用于食品中重要食源性致病菌的鉴定,分子分型,耐药,血清分型等。
展开全文
国内进展
2013年国家食品安全风险评估中心(CFSA)首次在国内采用全基因组测序技术处理新西兰恒天然婴儿配方食品污染可疑肉毒梭菌的食品安全应急事件,如图1所示。
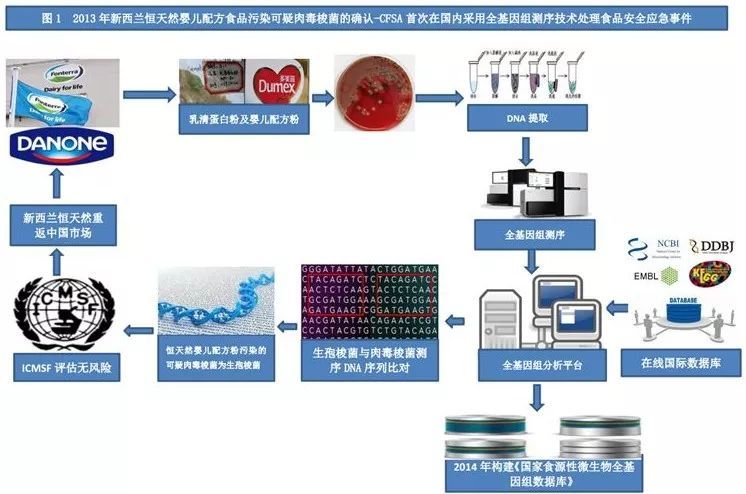
图1 2013年新西兰恒天然婴儿配方食品污染可疑肉毒梭菌的确认-全基因组测序技术在国内的首次应用
基于全基因测序技术已经成为国际上食源性疾病暴发病原溯源的金标准技术,2015年国家食品安全风险评估中心启动了《国家食源性致病微生物全基因组测序数据库》的构建工作,以应用于病因食品的快速准确识别,确保在技术上的国际话语权,技术流程如图2所示。

图2 国家食源性致病微生物全基因组数据库和溯源云平台构建
进入“十三五”以来,国内对微生物全基因组测序技术已经取得共识,需要迅速将该技术用于食源性致病菌鉴定,分型及溯源,方可在处理国家间微生物污染的食品安全事件中获得话语权。
《国家食源性致病微生物全基因组测序数据库》进展
1)数据库已收入1400余株食源性致病菌的全基因组序列数据
对国家污染物监测网络上报的菌株进行选择性的质量控制,筛选出具有代表性表性特征,如具有耐药性或致病性的菌株、持留株或具有生物膜形成能力的菌株进行全基因组测序。将下机原始数据、基因组序列数据及注释信息储存在本地数据库中。截止目前,数据库已收入1400余株食源性致病菌的全基因组序列数据,如图3所示。
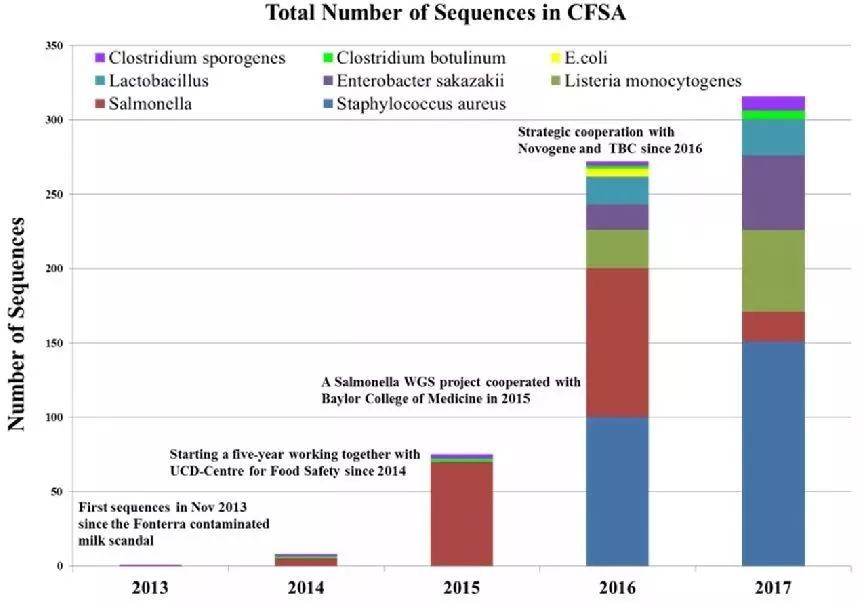
图3 食源性致病菌的测序数
2)全基因组测序数据分析平台
全基因组测序数据分析平台整合了包括原始数据处理、菌株鉴定、基因型特征分析和分子学溯源功能的4个不同功能模块。包括:
上一篇:FSU生物学家使用基因组数据库调查癌细胞
下一篇:机器学习揭示了构建人工蛋白质的方法